Acides Nucléiques
ADN Strong-Stop
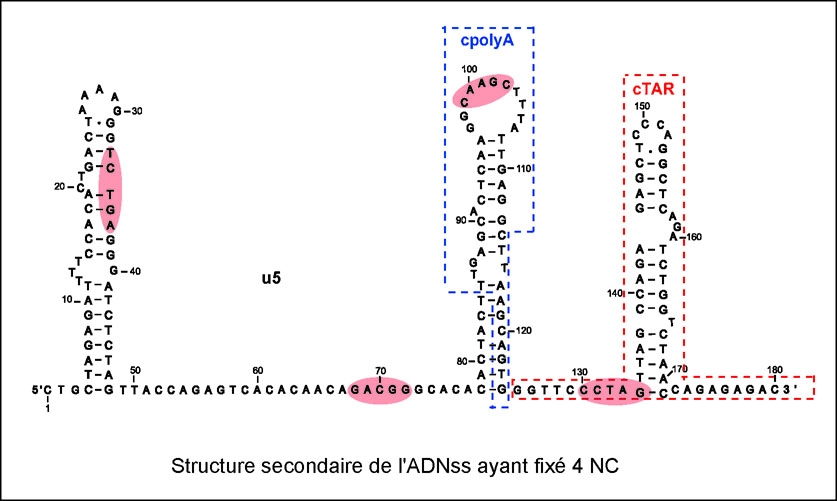
La réplication du virus de l’immunodéficience humaine de type 1 (VIH-1), agent causal du SIDA, dépend comme tous les rétrovirus de la transcription inverse de son ARN génomique (ARNg). Le 1er transfert de brin, une étape essentielle de la transcription inverse, se produit de l’extrémité 5’ de l’ARNg vers l’extrémité 3’ de l’ARNg au niveau des séquences R dupliquées. Il a lieu après la synthèse de l’ADN simple-brin (-) strong-stop (ADNss) par la transcriptase inverse (RT) du VIH-1. L’ADNss contient les régions complémentaires de R et U5. Au cours de la synthèse d'ADNss, les régions R et U5 de l'ARNg sont copiées par l’activité polymérase de la RT et sont dégradées par son activité RNase H. De ce fait, l'ADNss se désengage de sa matrice ARN située en 5' du génome et peut se replier avant de s'apparier via sa séquence r avec la séquence R qui lui est complémentaire et est située dans l’extrémité 3’ de l’ARNg. Le 1er transfert de brin repose donc en grande partie sur la formation d’un hybride ADN-ARN impliquant la région r de l’ADNss et la région R située dans l’extrémité 3’ de l’ARNg. L’extrémité 3’ de l’ARNg se replie en structures secondaires. Parmi celles-ci, les tiges-boucles TAR et poly(A) sont formées par la séquence R. Nous avons été les premiers à déterminer la structure secondaire de l’ADNss. La séquence r de l’ADNss forme deux structures en tige-boucle qui sont complémentaires des tiges-boucles ARN TAR et poly(A) et nommées pour cette raison ADN cTAR et ADN cpoly(A). L’appariement r-R nécessite donc une déstabilisation des structures secondaires de l’ADNss et de l’extrémité 3’ de l’ARNg. Les études in vitro suggèrent que la protéine de nucléocapside (NC) du VIH-1, qui possède une activité chaperonne des acides nucléiques, est impliquée dans cette déstabilisation. De plus, la NC facilite le 1er et le second transfert de brin dans des systèmes expérimentaux permettant de reconstituer in vitro ces processus. Il n’est pas possible d’étudier directement le rôle de la NC dans la transcription inverse ex vivo et par conséquent dans le 1er et le second transfert de brin.
Plusieurs études récentes supportent l’hypothèse que le désassemblage de la particule virale (décapsidation) est initié par la transcription inverse de l’ARNg. Des études sont en faveur d’un déclenchement de la décapsidation entre le 1er et le second transfert de brin. Toutefois, cela n’a pas été formellement démontré. A ce jour, les mécanismes moléculaires qui gouvernent les deux transferts de brin et la décapsidation ne sont que partiellement élucidés.
Notre recherche s’appuie sur nos résultats in vitro qui sont les seuls à avoir été obtenus avec l’ADNss entier. Plus précisément, elle a pour but de déterminer ex vivo si le 1er et le second transfert de brin sont altérés lorsque des tiges des structures secondaires de l’ADNss et de l’extrémité 3’ de l’ARNg sont stabilisées par des mutations ou lorsque les quatre sites forts NC sur l’ADNss sont supprimés par mutagénèse dirigée. Les objectifs de notre recherche sont de déterminer ex vivo : i) s’il existe une relation entre la structure secondaire de l’ADNss et les deux transferts de brin ; ii) si la NC est l’acteur principal qui facilite l’hybridation de l’ADNss avec l’extrémité 3’ de l’ARNg en déstabilisant ces deux molécules structurées, et si c’est le cas mieux comprendre comment la NC agit dans cette étape d’hybridation ; iii) si le second transfert de brin nécessite que les structures secondaires des régions r et u5 de l’ADNss soient déstabilisées par la NC pour pouvoir être copiées par la RT ; iv) si des mutations dans l’ADNss qui bloquent la transcription inverse au niveau du 1er ou du second transfert de brin ont un effet sur la décapsidation.